Cada día, médicos, enfermeras, fisioterapeutas, psicólogos, nutricionistas, etc. describen el estado de sus pacientes en forma de notas clínicas que se guardan en bases de datos de hospitales, consultorios y de atención primaria. Estainformación almacenada en forma de texto(información NO estructurada) representa el80 % de los datosde salud de los pacientes. Esto hace de los sistemas NLP una piedra angular en el aprovechamiento de los datos clínicos.
Para desarrollar sistemas de calidad de NLP clínico y para evitar las dificultades de acceso a datos clínicos reales, se han popularizado lascampañas de evaluación. Tienen carácter competitivo: sistemas procedentes de equipos de universidades e industria se enfrentan para conseguir el mejor rendimiento en una tarea concreta. Para ello, los organizadores preparan un conjunto de datos reales ya publicados, los anotan de forma consistente siguiendo guías de anotación, y, finalmente, los distribuyen entre los participantes, que los utilizarán para entrenar modelos predictivos que serán evaluados frente a un sistema de referencia. Como resultado, se liberan datos anotados y escenarios de evaluación, se comparan estrategias de manera objetiva y se generan publicaciones científicas al respecto.
La mayoría de campañas de NLP Clínico se llevan a cabo con datos en inglés. Sin embargo, en los últimos 5 años, desde el Barcelona Supercomputing Center hemos organizado más de 10 campañas basadas en datos en español. Estas campañas han generado un gran interés, con +11.000 visualizaciones y +3.000 descargas para los datos anotados liberados en las 3 tareas de 2020. Estas tareas contaron con participantes de más de 15 países, que publicaron 37 artículos científicos sobre el desarrollo de sistemas de NLP Clínico para textos en español.
Este año (2022), tenemos 4 campañas de evaluación de NLP Clínico en español en marcha y, ¡en todas ellas el proceso de registro está abierto! Os dejo aquí una pequeña descripción y el enlace a la web de cada una de ellas:
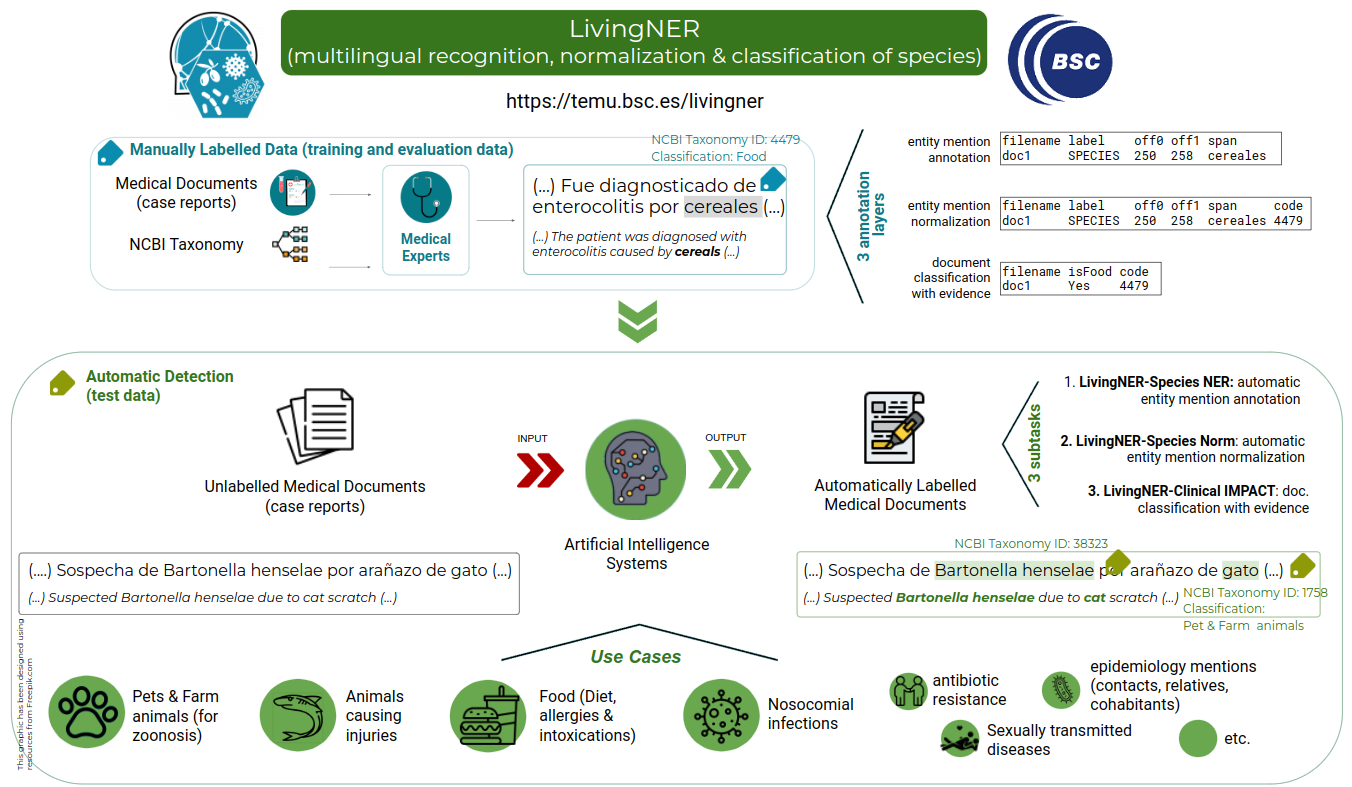
- LivingNER. La correcta detección de especies (patógenos, alimentos, animales, etc) en los informes médicos puede servir para aplicaciones tan diversas como la clasificación de alergias alimentarias o la detección de enfermedades nosocomiales. LivingNER pide a los participantes que creen sistemas para (1) reconocer menciones de especies; (2) normalizarlas aNCBI Taxonomy; y (3) detectar menciones relacionadas con animales de compañía, ataques de animales, alimentación e infecciones nosocomiales. Esto lo harán gracias a un corpus anotado manualmente por expertos clínicos.
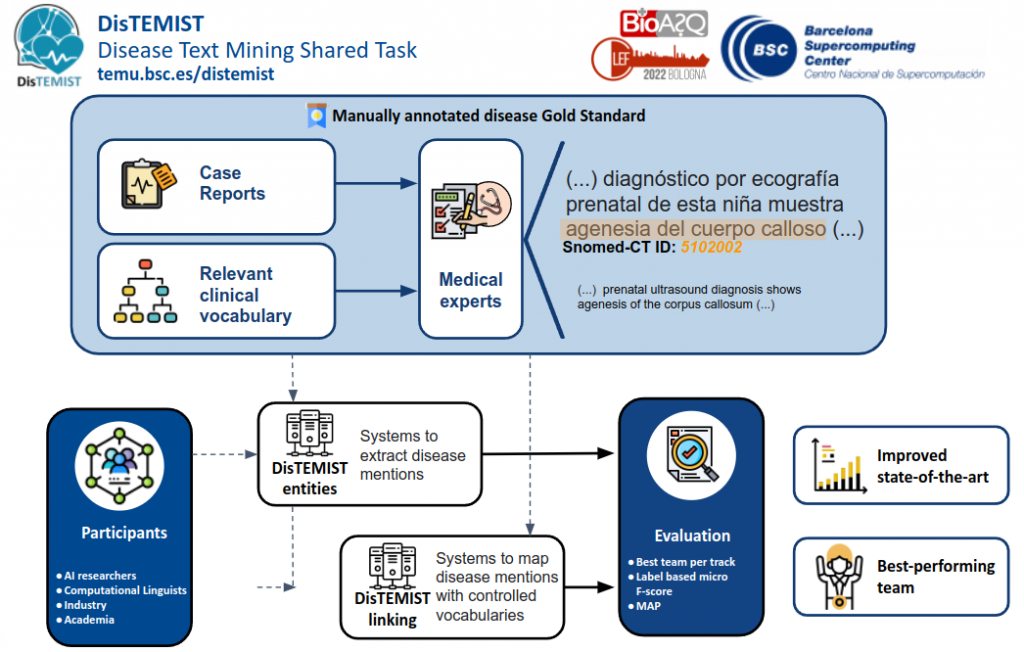
- DISTEMIST. Es evidente que las menciones de enfermedades son una pieza central dentro de toda la información contenida en los informes médicos. Sin embargo, DISTEMIST es la primera campaña enfocada específicamente en la detección y normalización de enfermedades en documentos clínicos en castellano. Los participantes deberán reconocer menciones de enfermedades y asignar a cada una de ellas un concepto de la terminologíaSnomed-CT. De nuevo, tienen a su disposición un corpus de documentos clínicos anotado manualmente por expertos.
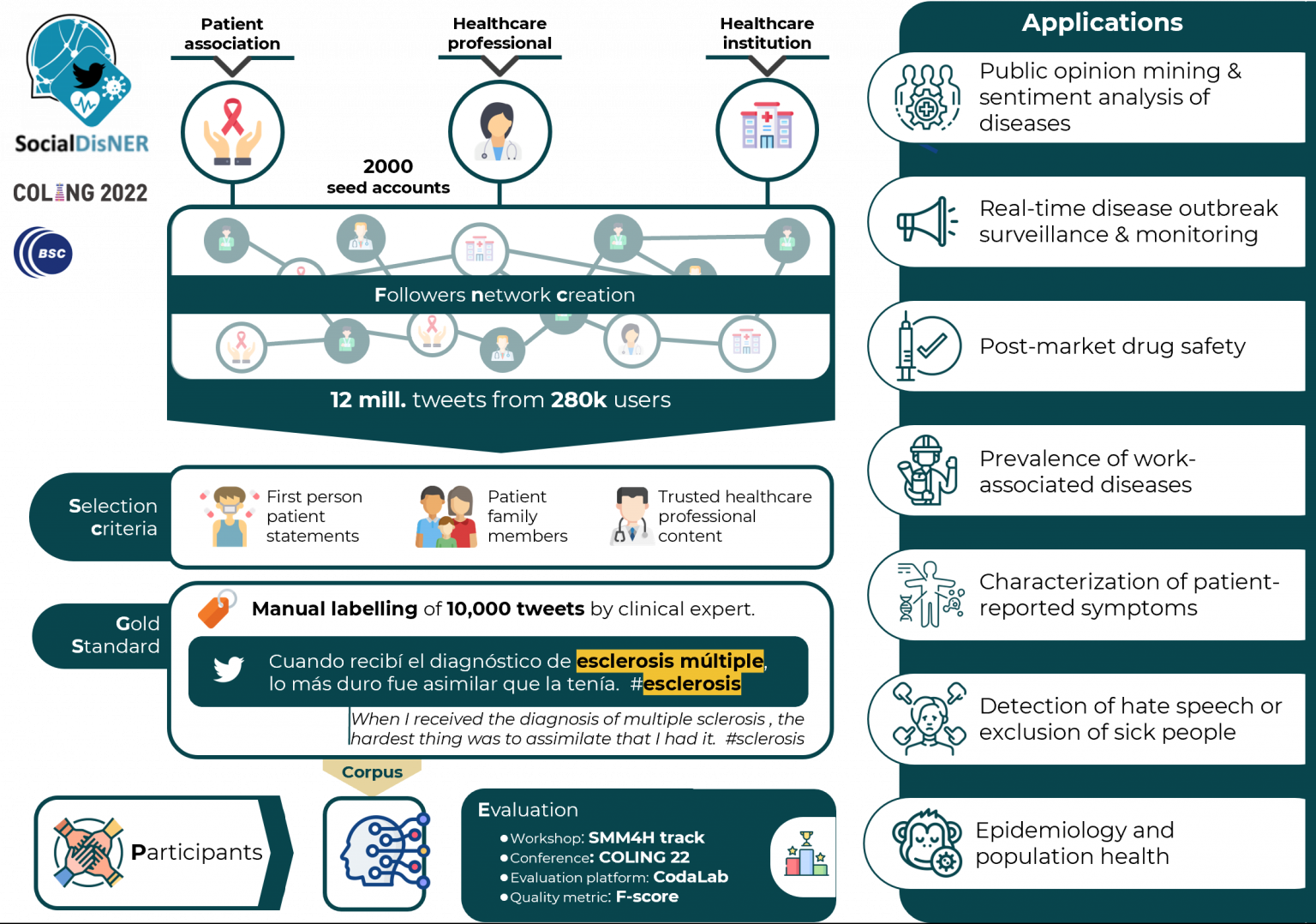
- SocialDisNER. Su objetivo es el desarrollo de sistemas que permitan estructurar la información de redes sociales para comprender mejor la percepción social de enfermedades de alta prevalencia, como cáncer y diabetes, y diagnósticos más complejos como la fibromialgia y los trastornos mentales. Para ello, los participantes crearán sistemas que permitan reconocer menciones de enfermedades en tweets escritos en español utilizando un corpus anotado manualmente por expertos médicos que incluye lenguaje informal y profesional.
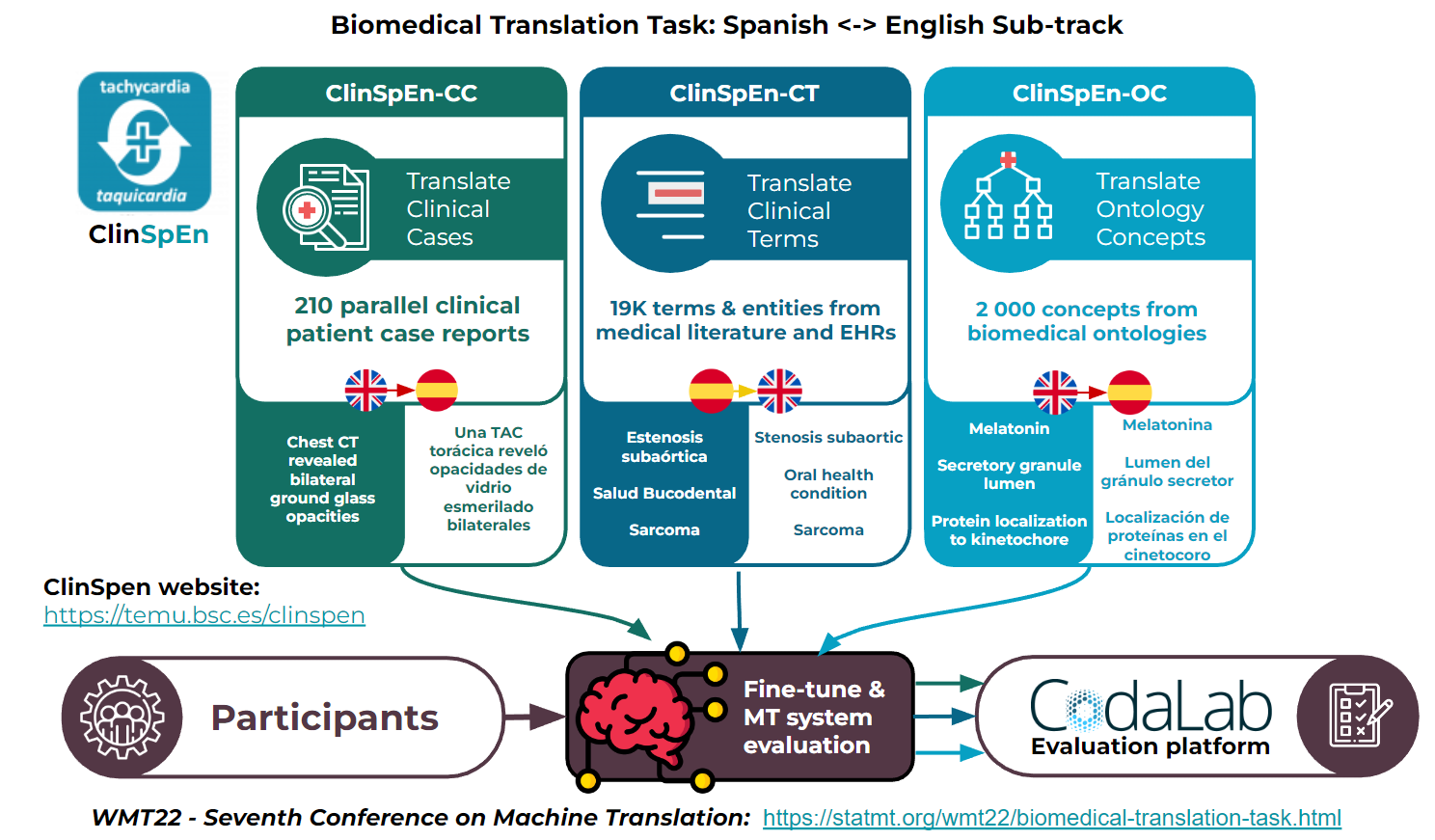
- ClinSpEn. Este año, los participantes en la tarea de traducción automáticaWMTdisponen de datos específicos para la traducción automática de recursos biomédicos en castellano. En concreto, hay planeada una tarea de traducción de casos clínicos, otra centrada en términos extraídos de informes médicos y una última sobre conceptos de ontologías biomédicas.
Estas 4 actividades han sido financiadas por elPlan de Tecnologías del Lenguaje.